library(tidyverse)7 Importer et exporter des données
R n’est pas prévu pour la saisie de données, mais il bénéficie de nombreuses fonctions et packages permettant l’import de données depuis un grand nombre de formats. Seuls les plus courants seront abordés ici.
Il est très vivement conseillé de travailler avec les projets de RStudio pour faciliter l’accès aux fichiers et pouvoir regrouper l’ensemble des éléments d’une analyse dans un dossier (voir Section 5.1).
Les projets permettent notamment de ne pas avoir à spécifier un chemin complet vers un fichier (sous Windows, quelque chose du genre C:\\Users\\toto\\Documents\\quanti\\projet\\data\\donnees.xls) mais un chemin relatif au dossier du projet (juste donnees.xls si le fichier se trouve à la racine du projet, data/donnees.xls s’il se trouve dans un sous-dossier data, etc.)
7.1 Import de fichiers textes
L’extension readr, qui fait partie du tidyverse, permet l’importation de fichiers texte, notamment au format CSV (Comma separated values), format standard pour l’échange de données tabulaires entre logiciels.
Cette extension fait partie du “coeur” du tidyverse, elle est donc automatiquement chargée avec :
Si votre fichier CSV suit un format CSV standard (c’est le cas s’il a été exporté depuis LibreOffice par exemple), avec des champs séparés par des virgules, vous pouvez utiliser la fonction read_csv en lui passant en argument le nom du fichier :
d <- read_csv("fichier.csv")Si votre fichier vient d’Excel, avec des valeurs séparées par des points virgule, utilisez la fonction read_csv2 :
d <- read_csv2("fichier.csv")Dans la même famille de fonction, read_tsv permet d’importer des fichiers dont les valeurs sont séparées par des tabulations, et read_delim des fichiers délimités par un séparateur indiqué en argument.
Chaque fonction dispose de plusieurs arguments, parmi lesquels :
col_namesindique si la première ligne contient le nom des colonnes (TRUEpar défaut)col_typespermet de spécifier manuellement le type des colonnes sireadrne les identifie pas correctementnaest un vecteur de chaînes de caractères indiquant les valeurs devant être considérées comme manquantes. Ce vecteur vautc("", "NA")par défaut
Il peut arriver, notamment sous Windows, que l’encodage des caractères accentués ne soit pas correct au moment de l’importation. On peut alors spécifier manuellement l’encodage du fichier importé à l’aide de l’option locale. Par exemple, si l’on est sous Mac ou Linux et que le fichier a été créé sous Windows, il est possible qu’il soit encodé au format iso-8859-1. On peut alors l’importer avec :
d <- read_csv("fichier.csv", locale = locale(encoding = "ISO-8859-1"))À l’inverse, si vous importez un fichier sous Windows et que les accents ne sont pas affichés correctement, il est sans doute encodé en UTF-8 :
d <- read_csv("fichier.csv", locale = locale(encoding = "UTF-8"))Pour plus d’informations sur ces fonctions, voir le site de l’extension readr.
À noter que si vous souhaitez importer des fichiers textes très volumineux le plus rapidement possible, la fonction fread de l’extension data.table est plus rapide que read_csv.
7.1.1 Interface interactive d’import de fichiers
RStudio propose une interface permettant d’importer un fichier de données de manière interactive. Pour y accéder, dans l’onglet Environment, cliquez sur le bouton Import Dataset :
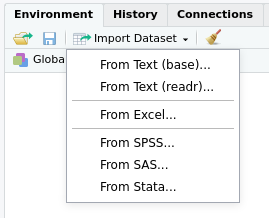
Sélectionnez From Text (readr)…. Une nouvelle fenêtre s’affiche :
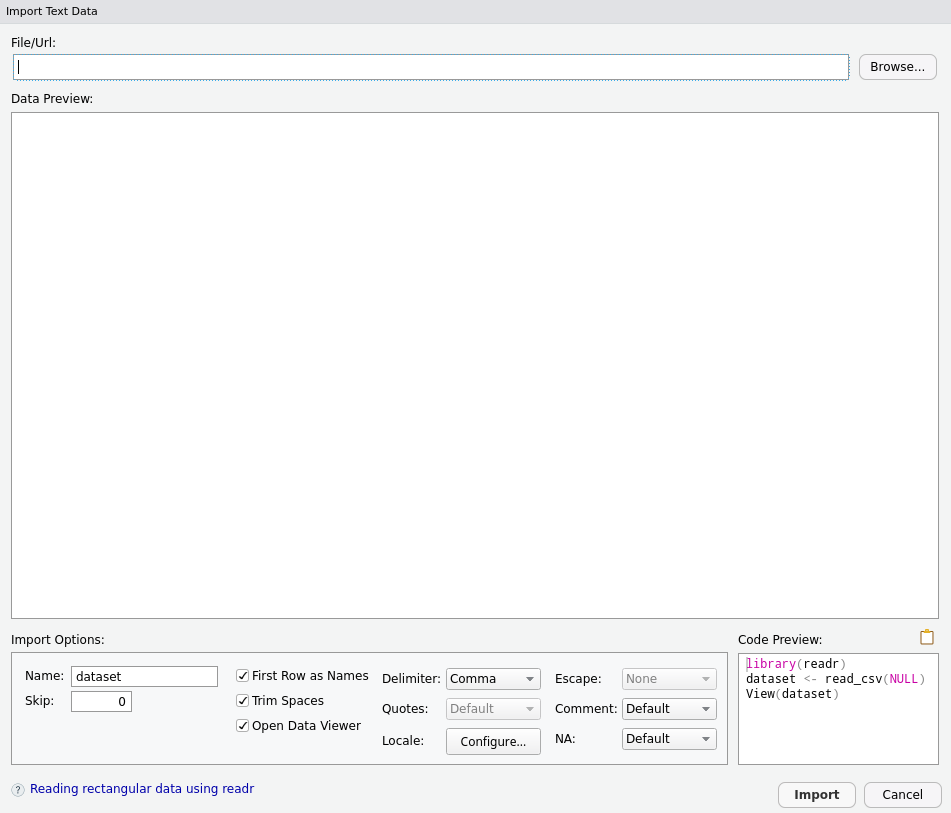
Il vous suffit d’indiquer le fichier à importer dans le champ File/URL tout en haut (vous pouvez même indiquer un lien vers un fichier distant via HTTP). Un aperçu s’ouvre dans la partie Data Preview et vous permet de vérifier si l’import est correct :
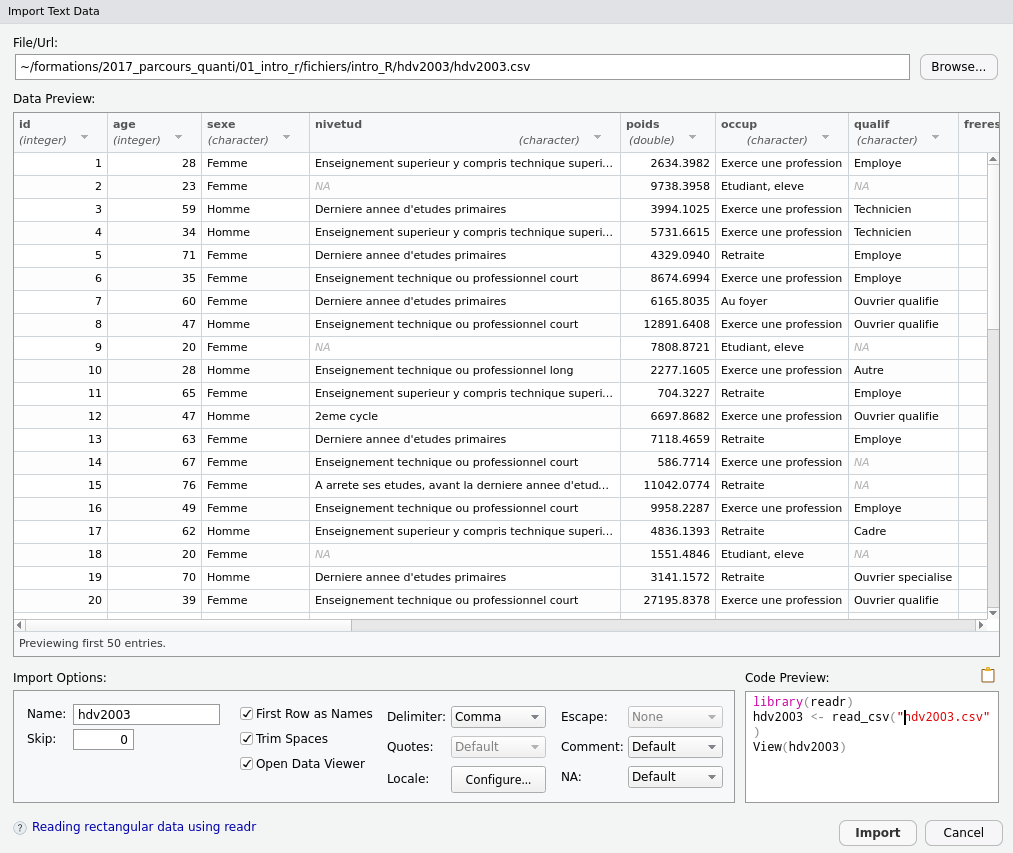
Vous pouvez modifier les options d’importation, changer le type des colonnes, etc. et l’aperçu se met à jour. De même, le code correspondant à l’importation du fichier avec les options sélectionnées est affiché dans la partie Code Preview.
Important : une fois que l’import semble correct, ne cliquez pas sur le bouton Import. À la place, sélectionnez le code généré et copiez-le (ou cliquez sur l’icône en forme de presse papier) et choisissez Cancel. Ensuite, collez le code dans votre script et exécutez-le (vous pouvez supprimer la ligne commençant par View).
Cette manière de faire permet “d’automatiser” l’importation des données, puisqu’à la prochaine ouverture du script vous aurez juste à exécuter le code en question, sans repasser par l’interface d’import.
7.2 Import depuis un fichier Excel
L’extension readxl, qui fait également partie du tidyverse, permet d’importer des données directement depuis un fichier au format xlsou xlsx.
Elle ne fait pas partie du “coeur” du tidyverse, il faut donc la charger explicitement avec :
library(readxl)On peut alors utiliser la fonction read_excel en lui spécifiant le nom du fichier :
d <- read_excel("fichier.xls")Il est possible de spécifier la feuille et la plage de cellules que l’on souhaite importer avec les arguments sheet et range :
d <- read_excel("fichier.xls", sheet = "Feuille2", range = "C1:F124")Comme pour l’import de fichiers texte, une interface interactive d’import de fichiers Excel est disponible dans RStudio dans l’onglet Environment. Pour y accéder, cliquez sur Import Dataset puis From Excel….
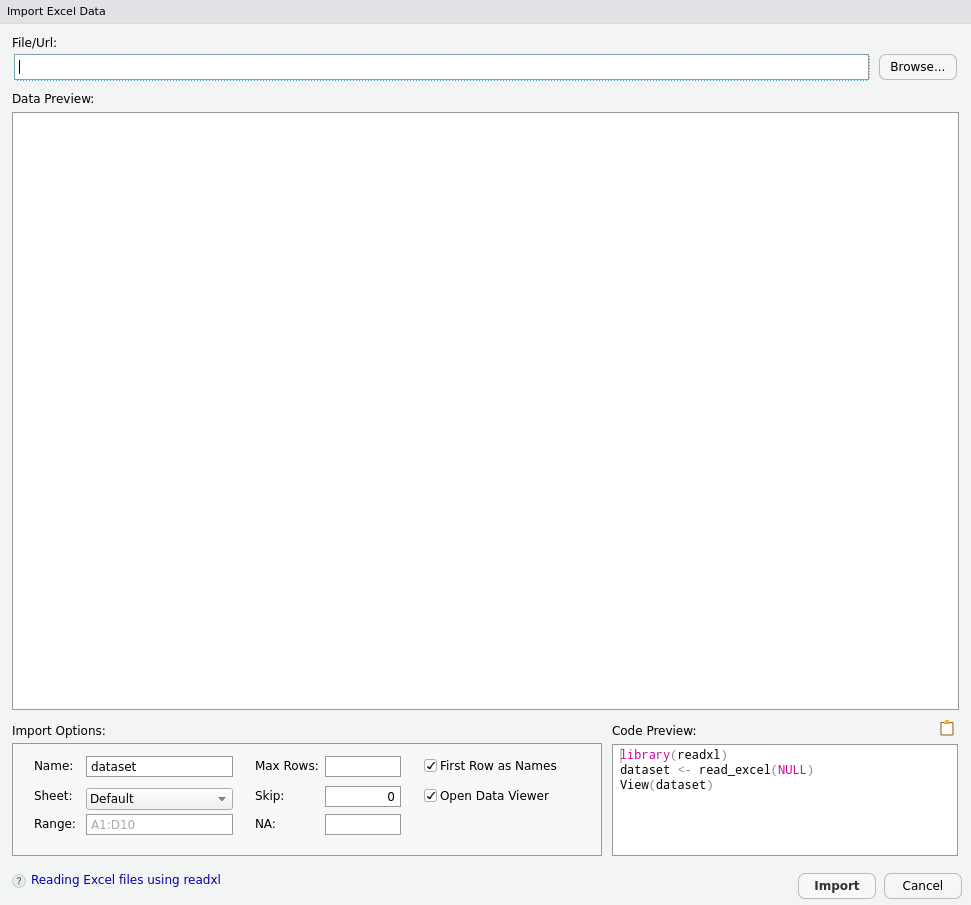
Spécifiez le chemin ou l’URL du fichier dans le premier champ, vérifiez l’import dans la partie Data Preview, modifiez si besoin les options d’importation, copiez le code d’importation généré dans la partie Code Preview et collez le dans votre script.
Pour plus d’informations, voir le site de l’extension readxl.
7.3 Import de fichiers SAS, SPSS et Stata
L’import de fichiers de données au format SAS, SPSS ou Stata se fait via les fonctions de l’extension haven.
Celle-ci fait partie du tidyverse, mais doit être chargée explicitement avec :
library(haven)- Pour les fichiers provenant de SAS, vous pouvez utiliser les fonctions
read_sasouread_xpt - Pour les fichiers provenant de SPSS, vous pouvez utiliser
read_savouread_por - Pour les fichiers provenant de Stata, utilisez
read_dta
Chaque fonction dispose de plusieurs options. Le plus simple est d’utiliser, là aussi l’interface interactive d’importation de données de RStudio : dans l’onglet Environment, sélectionnez Import Dataset puis From SPSS, From SAS ou From Stata. Indiquez le chemin ou l’url du fichier, réglez les options d’importation, puis copiez le code d’importation généré et collez le dans votre script.
Pour plus d’informations, voir le site de l’extension haven
7.4 Import de fichiers dBase
Le format dBase est encore utilisé, notamment par l’INSEE, pour la diffusion de données volumineuses.
Les fichiers au format dbf peuvent être importées à l’aide de la fonction read.dbf de l’extension foreign 1 :
library(foreign)
d <- read.dbf("fichier.dbf")La fonction read.dbf n’admet qu’un seul argument, as.is. Si as.is = FALSE (valeur par défaut), les chaînes de caractères sont automatiquement converties en factor à l’importation. Si as.is = TRUE, elles sont conservées telles quelles.
7.5 Connexion à des bases de données
7.5.1 Interfaçage via l’extension DBI
R est capable de s’interfacer avec différents systèmes de bases de données relationnelles, dont SQLite, MS SQL Server, PostgreSQL, MariaDB, etc.
Pour illustrer rapidement l’utilisation de bases de données, on va créer une base SQLite d’exemple à l’aide du code R suivant, qui copie la table du jeu de données mtcars dans une base de données bdd.sqlite :
library(DBI)
library(RSQLite)
con <- DBI::dbConnect(RSQLite::SQLite(), dbname = "resources/bdd.sqlite")
data(mtcars)
mtcars$name <- rownames(mtcars)
dbWriteTable(con, "mtcars", mtcars)
dbDisconnect(con)Si on souhaite se connecter à cette base de données par la suite, on peut utiliser l’extension DBI, qui propose une interface générique entre R et différents systèmes de bases de données. On doit aussi avoir installé et chargé l’extension spécifique à notre base, ici RSQLite. On commence par ouvrir une connexion à l’aide de la fonction dbConnect de DBI :
library(DBI)
library(RSQLite)
con <- DBI::dbConnect(RSQLite::SQLite(), dbname = "resources/bdd.sqlite")La connexion est stockée dans un objet con, qu’on va utiliser à chaque fois qu’on voudra interroger la base.
On peut vérifier la liste des tables présentes et les champs de ces tables avec dbListTables et dbListFields :
dbListTables(con)
#> [1] "mtcars"dbListFields(con, "mtcars")
#> [1] "mpg" "cyl" "disp" "hp" "drat" "wt" "qsec" "vs" "am" "gear"
#> [11] "carb" "name"On peut également lire le contenu d’une table dans un objet de notre environnement avec dbReadTable :
cars <- dbReadTable(con, "mtcars")On peut également envoyer une requête SQL directement à la base et récupérer le résultat :
dbGetQuery(con, "SELECT * FROM mtcars WHERE cyl = 4")
#> mpg cyl disp hp drat wt qsec vs am gear carb name
#> 1 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1 Datsun 710
#> 2 24.4 4 146.7 62 3.69 3.190 20.00 1 0 4 2 Merc 240D
#> 3 22.8 4 140.8 95 3.92 3.150 22.90 1 0 4 2 Merc 230
#> 4 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1 Fiat 128
#> 5 30.4 4 75.7 52 4.93 1.615 18.52 1 1 4 2 Honda Civic
#> 6 33.9 4 71.1 65 4.22 1.835 19.90 1 1 4 1 Toyota Corolla
#> 7 21.5 4 120.1 97 3.70 2.465 20.01 1 0 3 1 Toyota Corona
#> 8 27.3 4 79.0 66 4.08 1.935 18.90 1 1 4 1 Fiat X1-9
#> 9 26.0 4 120.3 91 4.43 2.140 16.70 0 1 5 2 Porsche 914-2
#> 10 30.4 4 95.1 113 3.77 1.513 16.90 1 1 5 2 Lotus Europa
#> 11 21.4 4 121.0 109 4.11 2.780 18.60 1 1 4 2 Volvo 142EEnfin, quand on a terminé, on peut se déconnecter à l’aide de dbDisconnect :
dbDisconnect(con)Ceci n’est évidemment qu’un tout petit aperçu des fonctionnalités de DBI.
7.5.2 Utilisation de dplyr et dbplyr
L’extension dplyr est dédiée à la manipulation de données, elle est présentée Chapitre 10. En installant l’extension complémentaire dbplyr, on peut utiliser dplyr directement sur une connection à une base de données générée par DBI :
library(DBI)
library(RSQLite)
library(dplyr)
con <- DBI::dbConnect(RSQLite::SQLite(), dbname = "resources/bdd.sqlite")La fonction tbl notamment permet de créer un nouvel objet qui représente une table de la base de données :
cars_tbl <- tbl(con, "mtcars")Ici l’objet cars_tbl n’est pas un tableau de données, c’est juste un objet permettant d’interroger la table de notre base de données.
On peut utiliser cet objet avec les verbes de dplyr :
cars_tbl %>%
filter(cyl == 4) %>%
select(name, mpg, cyl)
#> # Source: SQL [?? x 3]
#> # Database: sqlite 3.47.1 [/home/runner/work/tidyverse/tidyverse/resources/bdd.sqlite]
#> name mpg cyl
#> <chr> <dbl> <dbl>
#> 1 Datsun 710 22.8 4
#> 2 Merc 240D 24.4 4
#> 3 Merc 230 22.8 4
#> 4 Fiat 128 32.4 4
#> 5 Honda Civic 30.4 4
#> 6 Toyota Corolla 33.9 4
#> 7 Toyota Corona 21.5 4
#> 8 Fiat X1-9 27.3 4
#> 9 Porsche 914-2 26 4
#> 10 Lotus Europa 30.4 4
#> # ℹ more rowsdbplyr s’occupe, de manière transparente, de transformer les instructions dplyr en requête SQL, d’interroger la base de données et de renvoyer le résultat. De plus, tout est fait pour qu’un minimum d’opérations sur la base, parfois coûteuses en temps de calcul, ne soient effectuées.
Il est possible de modifier des objets de type tbl, par exemple avec mutate :
cars_tbl <- cars_tbl %>% mutate(type = "voiture")
Dans ce cas la nouvelle colonne type est bien créée et on peut y accéder par la suite. Mais cette création se fait dans une table temporaire : elle n’existe que le temps de la connexion à la base de données. À la prochaine connexion, cette nouvelle colonne n’apparaîtra pas dans la table.
Bien souvent on utilisera une base de données quand les données sont trop volumineuses pour être gérées par un ordinateur de bureau. Mais si les données ne sont pas trop importantes, il sera en général plus rapide de récupérer l’intégralité de la table dans notre session R pour pouvoir la manipuler comme les tableaux de données habituels. Ceci se fait grâce à la fonction collect de dplyr :
cars <- cars_tbl %>% collectIci, cars est bien un tableau de données classique, copie de la table de la base au moment du collect.
Et dans tous les cas, on n’oubliera pas de se déconnecter avec :
dbDisconnect(con)7.5.3 Ressources
Pour plus d’informations, voir la documentation très complète (en anglais) proposée par RStudio.
Par ailleurs, depuis la version 1.1, RStudio facilite la connexion à certaines bases de données grâce à l’onglet Connections. Pour plus d’informations on pourra se référer à l’article (en anglais) Using RStudio Connections.
7.6 Export de données
7.6.1 Export de tableaux de données
On peut avoir besoin d’exporter un tableau de données dans R vers un fichier dans différents formats. La plupart des fonctions d’import disposent d’un équivalent permettant l’export de données. On citera notamment :
write_csv,write_csv2,write_tsvpermettent d’enregistrer un data frame ou un tibble dans un fichier au format texte délimitéwrite_saspermet d’exporter au format SASwrite_savpermet d’exporter au format SPSSwrite_dtapermet d’exporter au format Stata
Il n’existe par contre pas de fonctions permettant d’enregistrer directement au format xls ou xlsx. On peut dans ce cas passer par un fichier CSV.
Ces fonctions sont utiles si on souhaite diffuser des données à quelqu’un d’autre, ou entre deux logiciels.
Si vous travaillez sur des données de grandes dimensions, les formats texte peuvent être lents à exporter et importer. Dans ce cas, d’autres extensions comme arrow ou fst peuvent être utiles : elles permettent d’enregistrer des data frames dans des formats plus rapides. Les formats proposés par arrow permettent en outre l’échange de données tabulaires avec d’autres langages de programmation comme Python ou JavaScript.
7.6.2 Sauvegarder des objets
Une autre manière de sauvegarder des données est de les enregistrer au format RData. Ce format propre à R est compact, rapide, et permet d’enregistrer plusieurs objets R, quel que soit leur type, dans un même fichier.
Pour enregistrer des objets, il suffit d’utiliser la fonction save et de lui fournir la liste des objets à sauvegarder et le nom du fichier :
save(d, rp2018, tab, file = "fichier.RData")Pour charger des objets préalablement enregistrés, utiliser load :
load("fichier.RData")Les objets d, rp2018 et tab devraient alors apparaître dans votre environnement.
Attention, quand on utilise load, les objets chargés sont importés directement dans l’environnement en cours avec leur nom d’origine. Si d’autres objets du même nom existent déjà, ils sont écrasés sans avertissement.
Une alternative est d’utiliser les fonctions saveRDS et readRDS, qui permettent d’enregistrer un unique objet, et de le charger dans notre session avec le nom que l’on souhaite.
saveRDS(rp2018, "fichier.rds")
df <- readRDS("fichier.rds")foreignest une extension installée de base avec R, vous n’avez pas besoin de l’installer, il vous suffit de la charger aveclibrary.↩︎